【文献分享】MaskGraphene:一种用于多层、多条件空间转录组学的可解释联合表示的高级框架
文章目录
- 介绍
- 代码
- 参考
介绍
空间转录组学(ST)领域的最新进展强调了需要整合多个切片以进行联合分析的必要性。一个关键挑战在于生成具有可解释性的嵌入,这些嵌入既能保留空间几何结构,又能纠正批次效应。我们提出了 MaskGraphene,这是一种通过掩码自监督学习、三元组损失和基于簇的局部对齐来整合 ST 数据的图神经网络。通过在切片之间建立间接的“软链接”和直接的“硬链接”,MaskGraphene 生成具有高几何保真度的联合嵌入。与八种方法的基准测试表明,其具有更优的对齐效果和可解释性。MaskGraphene 增强了下游应用,包括领域识别、轨迹重建、生物标志物发现和大脑层图谱绘制,从而实现了稳健的 ST 整合和生物学洞察力。

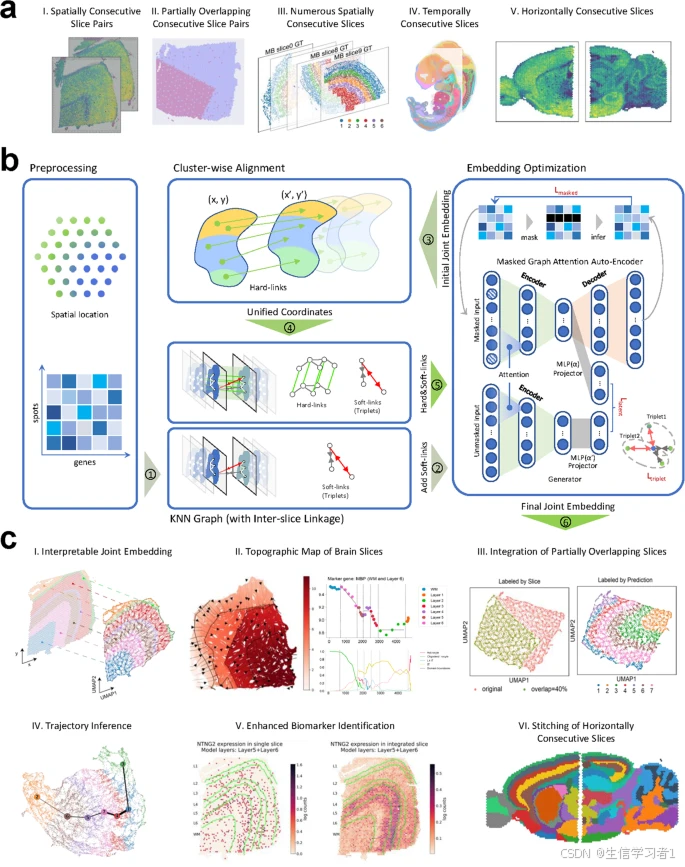
MaskGraphene 工作流程。a MaskGraphene 所解决的空间转录组学数据整合场景的示意图,包括空间上连续的切片对(I)、模拟部分重叠的连续切片对(II)、众多空间上连续的切片(III)、时间上连续的切片(IV)以及水平上连续的切片(V)。b MaskGraphene 的工作流程:预处理步骤对空间坐标和基因表达数据进行组织。通过基于聚类的局部比对构建“硬链接”以及使用三元组对比学习建立“软链接”来建立切片间的关联。嵌入优化利用掩码图自编码器生成批校正的联合嵌入,通过优化掩码自监督损失()和三元组损失来实现。主要方法的步骤 1 - 5 用编号圆圈表示。c 应用和评估(第 6 步):(I)可解释的联合嵌入捕捉原始几何结构。(II)具有等深度分析的脑切片地形图揭示了皮质层间基因表达梯度。(III)使用模拟数据进行验证表明对部分重叠切片的整合具有鲁棒性。(四)轨迹推断揭示了线性关联的发展趋势。(五)胚胎组织结构的对齐与整合有助于生物标志物的识别。(六)水平连续切片的拼接可重建空间上连贯的区域。
生物体内复杂的生命活动过程依赖于不同细胞类型的多样性和专业化,每个细胞类型都经过精心设计以完成特定的功能。要揭示疾病病理和组织功能的复杂性,理解相邻细胞和远处细胞之间的联系与相互作用至关重要,因为细胞的行为深受其微环境的影响[1]。尽管单细胞 RNA 测序(scRNA-seq)极大地提高了我们以单细胞分辨率分析基因表达的能力,但其缺乏空间背景限制了我们对细胞在其天然微环境中的生态位的理解。这种局限性阻碍了我们对细胞间相互作用、组织结构以及空间调控的功能动态等关键过程的理解,而这些对于解析生物系统来说都是至关重要的[2, 3]。
空间转录组学(ST)的进展通过同时测量组织切片内的 mRNA 表达和空间坐标,实现了转录组分析与空间背景之间的衔接[4]。这些技术极大地提升了我们探索异质组织复杂转录景观的能力[5]。现有的方法大致可分为基于成像的技术,如 smFISH、STARmap、MERFISH、seqFISH 和 seqFISH+,它们具有高空间分辨率;以及基于测序的技术,包括 Slide-seq、Slide-seqV2、10x Visium、HDST 和 Stereo-seq,这些技术能提供具有不同分辨率的可扩展数据[11,12,13,14,15]。尽管这些进展取得了显著成果,但在不同实验条件或技术下生成的 ST 数据集的综合分析仍是一个重大挑战[16]。
传统的单层 ST 数据分析主要致力于揭示单个组织切片内的空间域分布[17,18,19]。近年来,人们越来越认识到对来自不同来源(包括不同样本、生物条件、技术平台和发育阶段)的 ST 数据进行整合和比较分析的价值[20]。整合分析能提供对空间组织结构更全面的理解,从而有助于更深入地探究复杂的空间组织,并利用更多信息进行更可靠的分析。然而,ST 数据容易受到批次效应的影响,这可能会掩盖真实的生物信号并使数据解读变得复杂。因此,开发一个精心设计的多层 ST 数据整合框架,能够同时对不同层的信号进行建模和协调,并减轻批次效应的影响,是一个紧迫且关键的需求。
代码
https://github.com/maiziezhoulab/MaskGraphene
参考
- MaskGraphene: an advanced framework for interpretable joint representation for multi-slice, multi-condition spatial transcriptomics
- https://github.com/maiziezhoulab/MaskGraphene
