生物信息学 (101计划核心教程)Chapter4
第四章 生物信息资源
4.1 生物数据库简介
生物数据库是生命科学发展不可或缺的信息资源,也是合成生物学、生物医药与生物技术产业赖以发展的基础。生物数据库的建设、维护与安全关系人口健康、生物安全和可持续发展,是国家重要战略资源。
生物数据库类型种类繁多。根据存储数据内容的不同,可以分为DNA数据库、RNA数据库、蛋白质数据库、物种特异性数据库、其他数据库等。或基于数据获取的规模和复杂性程度,可细分为一级数据库和二级数据库,或者用户针对性更强的专用数据库等。
DNA数据库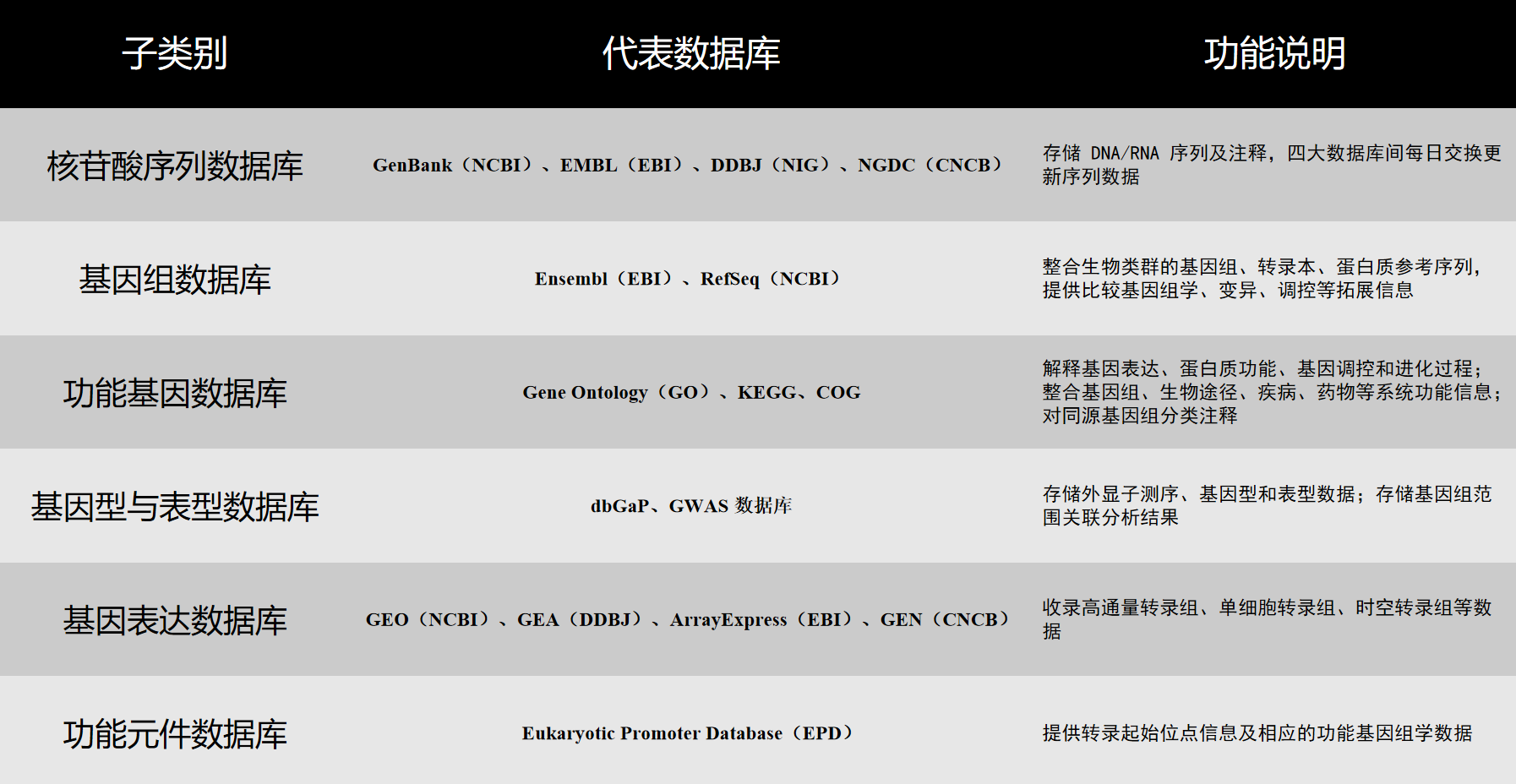
RNA数据库
蛋白质数据库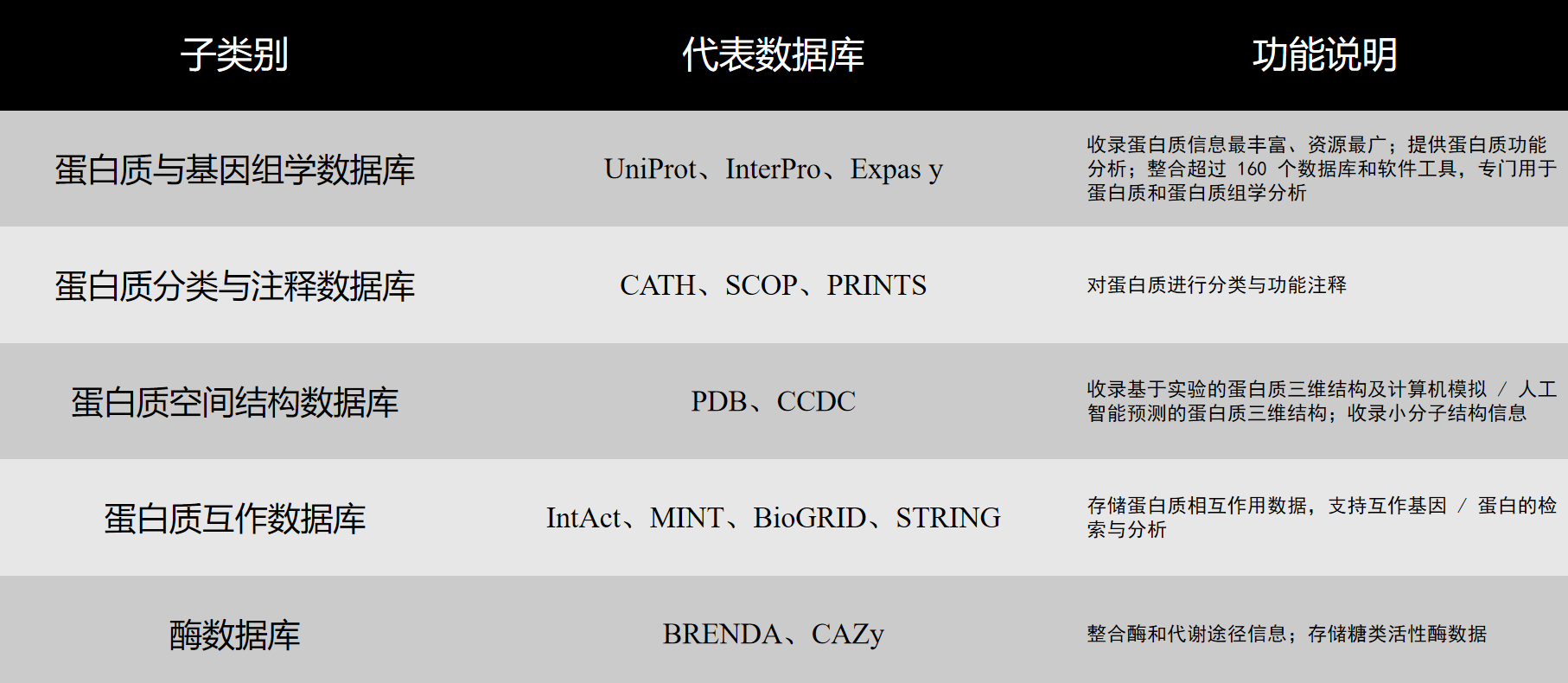
生物数据库期刊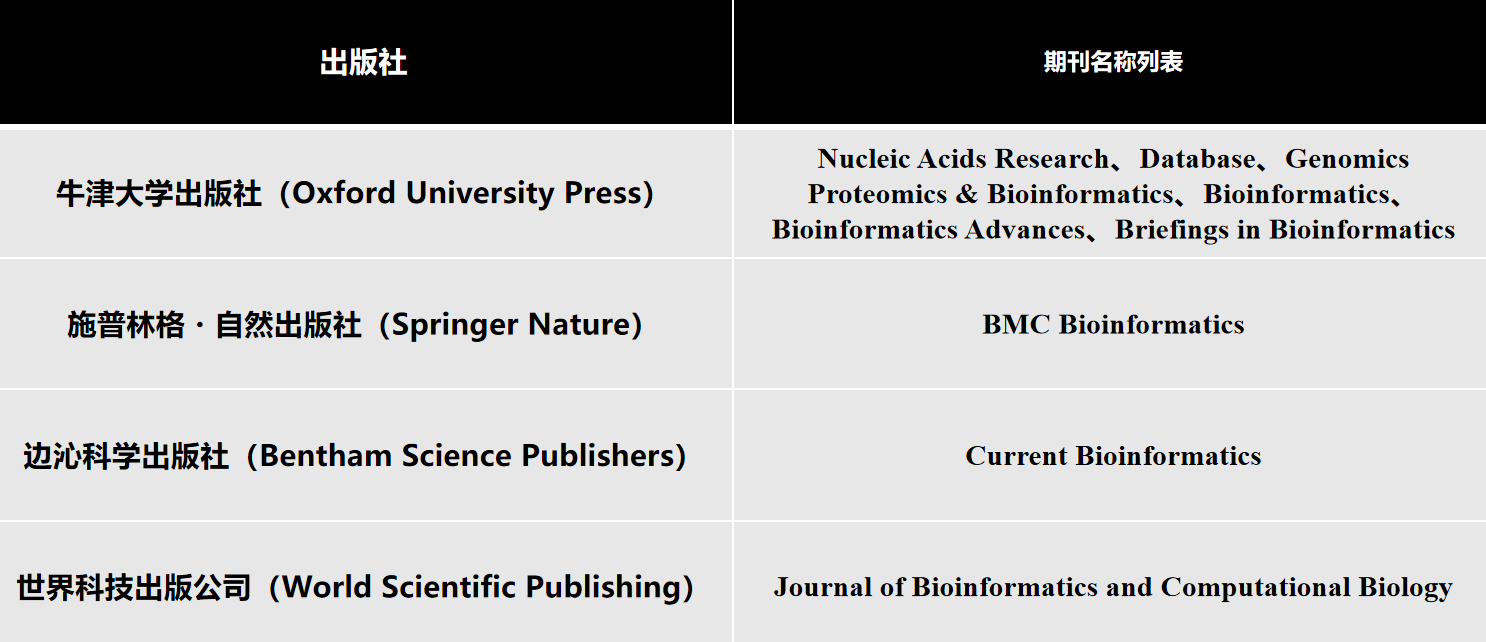
4.2国际主要数据中心
随着生命科学蓬勃发展,生物信息数据体量呈指数级递增,组学数据资源的收集管理已成为生物信息研究领域的基础需求。建立数据资源库和生物信息数据中心,是能够最有效地管理和使用海量生物大数据的有效途径。
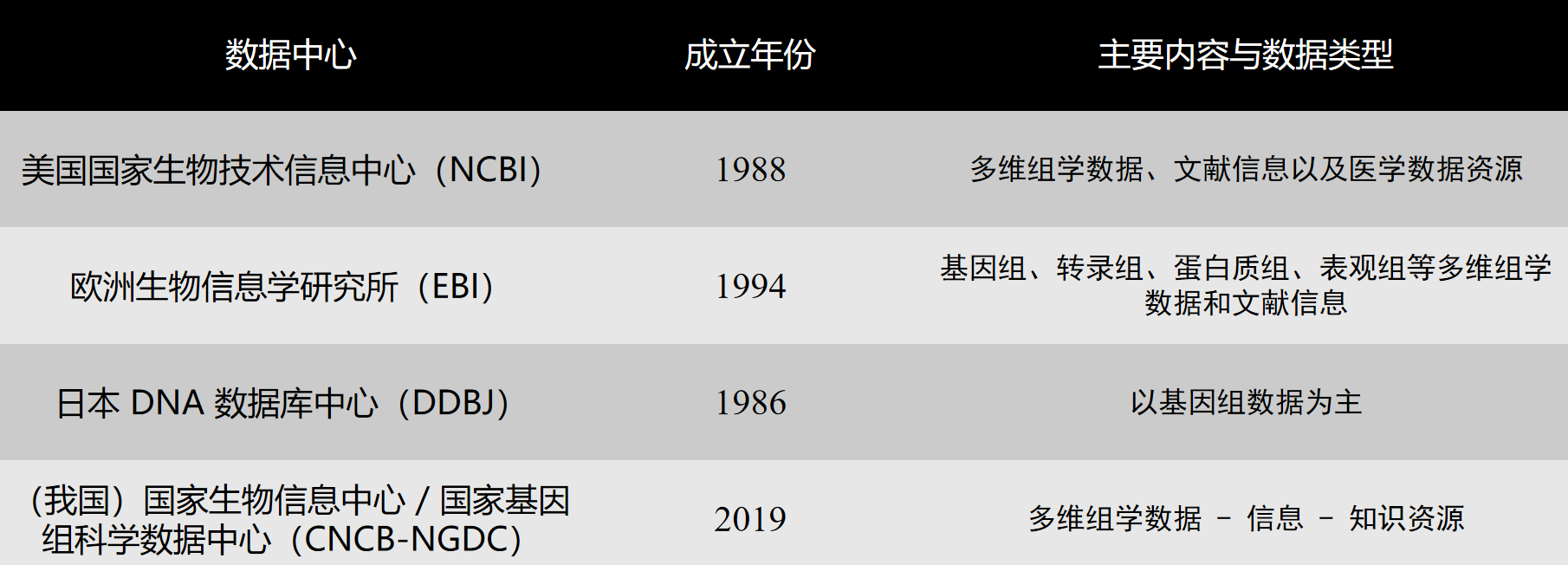
中国国家生物信息中心
CNCB-NGDC多组学数据库资源体系建设情况(见书中下图)
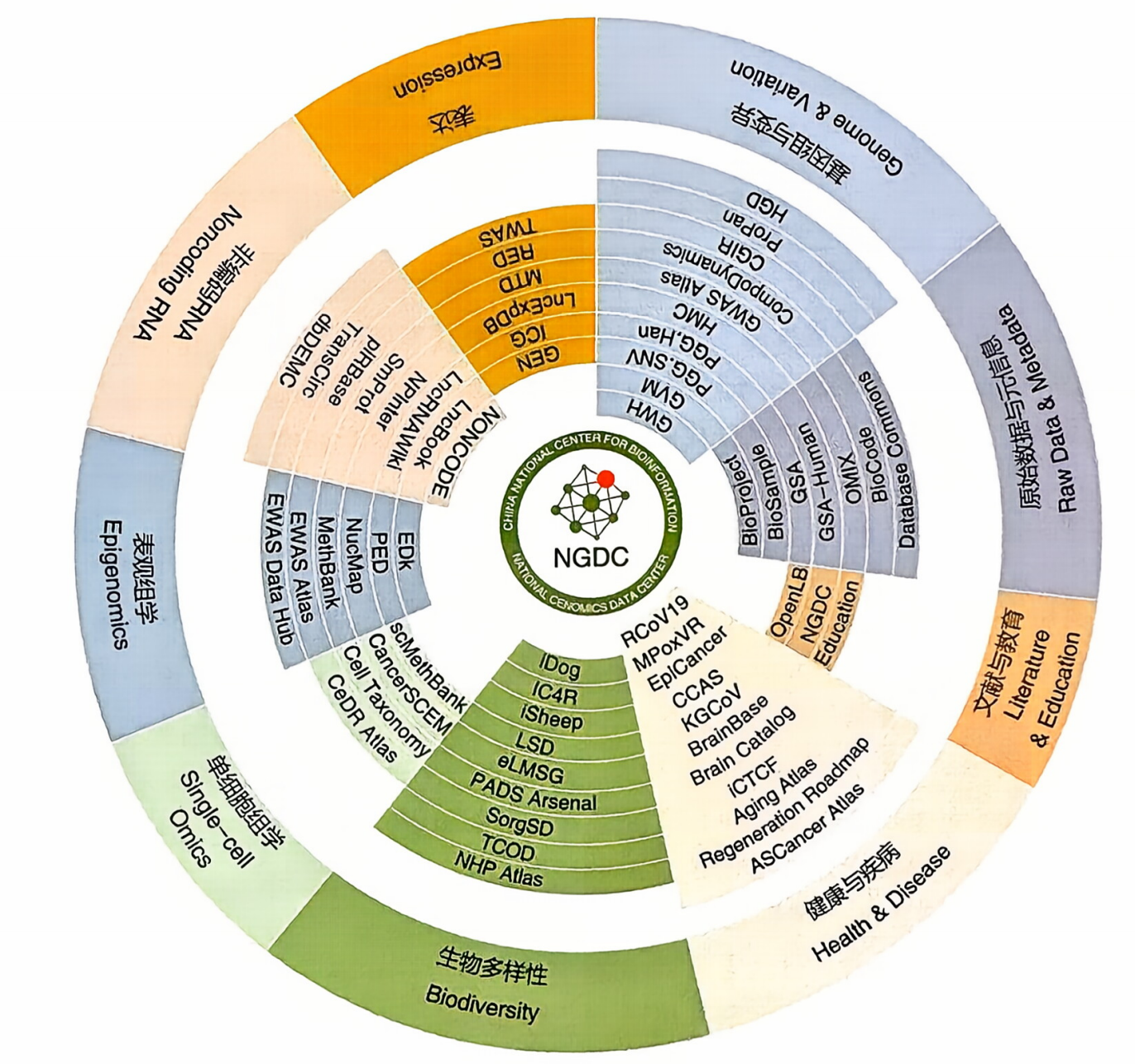
CNCB-NGDC的代表性资源

虽然我国的各大科研院所和高校已经建立了类型丰富的生物数据库,但与领域内国际顶尖生物数据库相比,在基础设施、数据汇交管理、好用易用性等方面仍存在差距,尤其在数据库的权威性、完整性、多维度、使用界面友好度、数据上传与下载快捷性、不同数据库智能关联性、人工智能辅助应用等方面存在较大差距。
美国国家生物技术信息中心
NCBI的主要目标是为科学家、医组学的广泛资源和工具,其代表性资源(可见下图)
现今,美国国家生物技术信息中心主要针对的是生物技术和生物医学领域的数据,致力于创立自动化系统用于储存和分析相关数据和知识,促进科研和医疗团体对这些数据库进行数据获取与使用,协调国内外生物数据信息的收集与整合。
欧洲生物信息学研究所
EMBL-EBI的代表性资源

这些资源和服务使得研究人员能够更好地利用生物信息学和基因组学的方法来解决生命科学中的问题
日本 DNA 数据库中心
DDBJ Sequence Read Archive(DRA) 2008 归档测序原始数据和比对信息的公共存储库
DDBJ(DDBJ) 2008 归档组装 / 注释的核苷酸序列
Japanese Genotype-Phenotype Archive(JGA) 2013 归档人类个体水平的基因型和表型信息,并提供数据的受控访问
NBDC Human Database(NHA) 2016 归档人类相关的基因组、遗传信息、临床信息、影像等数据,并提供数据的公开 / 受控访问
Genomie Expression Archive(GEA) 2018 归档芯片和高通量测序实验中的功能基因组学数据
MetaboBank(MetaboBank) 2020 归档从质谱、核磁共振和成像质谱获得的代谢组学数据的公共存储库
4.3代表性生物数据库
快速回顾一下按照4.1的分类的一些代表性生物数据库(见书中梳理如下),并对其中序列数据库、结构数据库以及互作数据库进行梳理:







4.3.1 序列数据库
在生物信息学研究中,序列数据库是连接 “核酸 / 蛋白质序列” 与 “生物学功能” 的核心桥梁。从基因组注释到蛋白质功能解析,一系列专业数据库构成了研究的 “基础设施”。
1. Ensembl:多物种基因组注释与可视化的 “百科全书”
Ensembl 是综合性生物信息学研究项目,最初聚焦脊椎动物基因组,后拓展至线虫、酵母、拟南芥、水稻等模式生物。随着测序技术发展,其收录的基因组数据持续扩容,功能也从单纯的序列注释拓展至比较基因组学、变异与调控注释等方向。
核心价值:
- 基因组注释的 “全景图”:提供基因结构、转录本变体、调控元件等详细注释;
- 比较基因组学工具:支持多物种基因组序列比对,揭示物种进化关系与功能保守性;
- 基因组可视化标杆:其基因组浏览器是展示基因组结构、变异位点、基因表达模式的 “黄金工具”。

3. GO 数据库:基因功能注释的 “通用语言”
基因本体(Gene Ontology, GO)数据库旨在建立跨物种的基因 / 蛋白功能语义标准,解决生物领域术语不统一导致的数据交流障碍。它从三个维度定义功能:
- 分子功能(MF):基因产物的分子级活动(如 “催化”“结合”);
- 细胞成分(CC):基因产物在细胞中的定位(如 “细胞核”“线粒体”);
- 生物过程(BP):基因参与的生物学事件(如 “细胞周期”“免疫应答”)。
GO 通过 “term” 定义功能标签,标签间以is_a、part_of、regulates等关系构建有向无环网络,形成功能层级结构(如书中下图)。
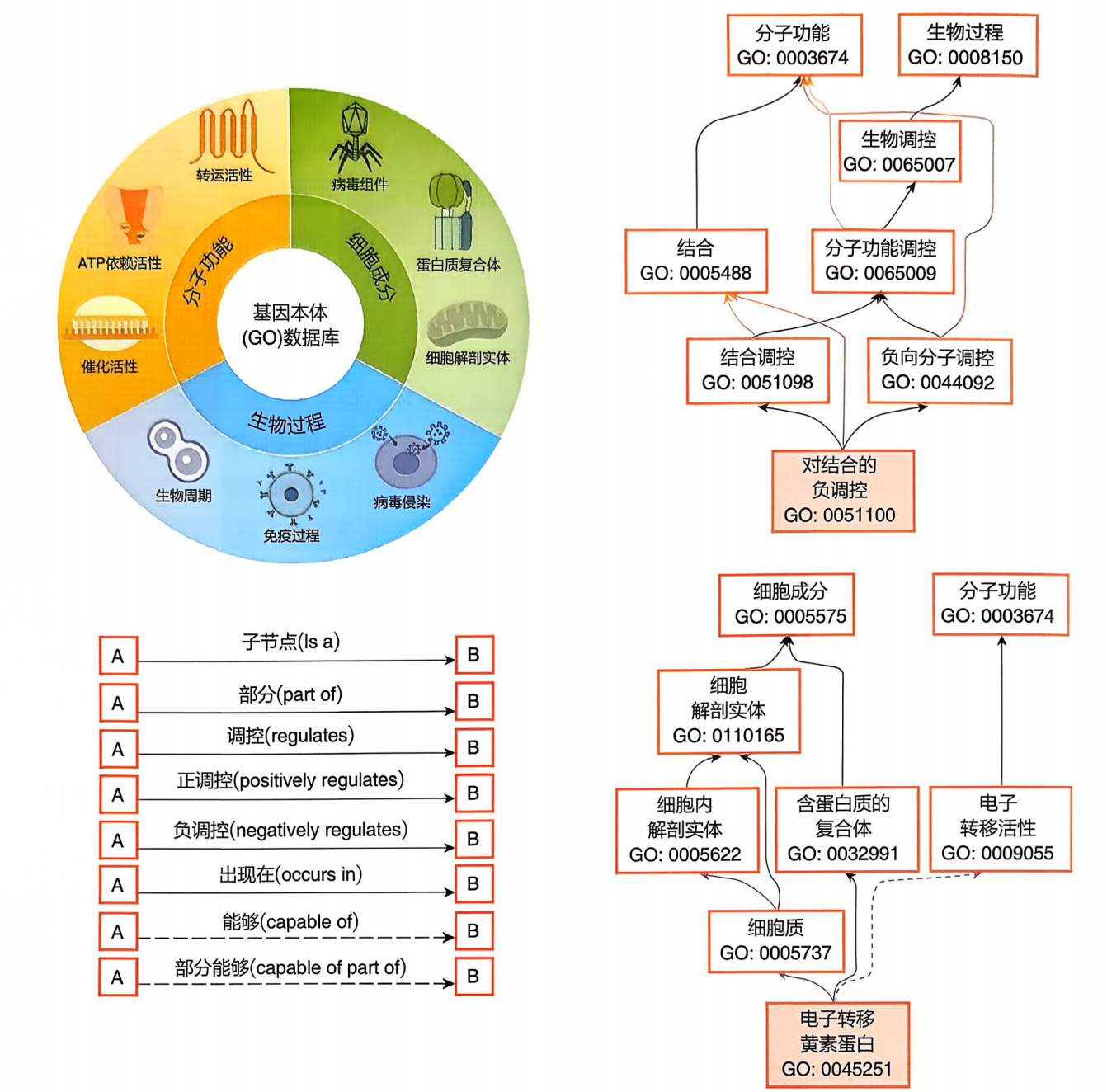
核心价值:
- 功能注释的 “翻译器”:让不同物种的基因功能具有统一表述(如人类 “TP53” 与小鼠 “Trp53” 可通过 GO 术语关联);
- 功能富集分析的 “引擎”:通过统计基因集的 GO 术语分布,揭示其核心生物学功能(如肿瘤组织中 “细胞增殖” 相关 GO 术语显著富集);
- 多组学数据的 “粘合剂”:整合基因组、转录组、蛋白质组数据,解析 “序列 - 功能 - 表型” 的关联。

5. 蛋白质结构域与无序区域:专精化研究的 “利器”
除了上述通用数据库,还有一批聚焦蛋白质特定特征的专精数据库,助力细分领域研究:
PRINTS 基于蛋白质的保守结构域和模体,通过多序列比对提取 “高保守序列片段”,形成蛋白质家族的 “指纹”。可应用于蛋白质家族识别、结构域功能预测(如通过 PRINTS 判断某蛋白是否属于 “激酶家族”)。
蛋白质无序区域是缺乏规则三维结构的序列片段,却在信号转导、分子识别、蛋白互作中发挥关键作用:
- DisProt:专注记录蛋白质无序区域的序列与功能;
- MobiDB:进一步区分 “固有无序区域” 与 “可折叠区域”,提供实验与计算数据(如序列长度、结构域界限、疾病关联)。
4.3.2 结构数据库
在生物信息学领域,结构数据库是解析生物大分子(核酸、蛋白质)三维结构与功能关系的核心资源。从实验测定的结构到 AI 预测的模型,从结构分类到功能域注释,一系列专业数据库构成了 “结构 - 功能 - 应用” 的研究闭环。
1. 核酸结构数据库:解析核酸构象与调控的 “透视镜”
核酸(DNA、RNA)的三维结构直接决定其功能(如基因调控、催化活性),以下数据库聚焦核酸结构研究:
核酸数据库(Nucleic Acid Database, NDB)
- 功能:存储 DNA、RNA 的三维结构信息,整合生物分子结晶学和核磁共振数据。
- 价值:助力研究核酸的构象变化、分子间相互作用(如 DNA - 蛋白质结合、RNA 折叠)。
- 应用场景:解析基因启动子的三维构象对转录的调控机制、研究 RNA 病毒的基因组结构与侵染性的关联。
核小体数据库(Nucleome Data Bank, NDB)
- 功能:专注染色质的空间结构、基因组亚结构及基因表达调控相关信息。
- 价值:揭示染色质三维组织与基因表达的关系(如增强子与启动子的远距离互作)。
- 应用场景:研究肿瘤细胞中染色质结构异常对癌基因激活的影响、解析表观遗传修饰如何通过染色质结构调控基因表达。
2. 蛋白质结构与功能域数据库:从序列到结构的 “解码器”
蛋白质的结构域、功能位点是其执行生物学功能的核心单元,以下数据库专注于蛋白质结构与功能的关联分析:
InterPro:蛋白质功能注释的 “超级整合平台”
- 功能:整合多个蛋白质数据库(Coils、Pfam、PRINTS 等),提供蛋白质家族归纳、结构域预测、功能位点注释。
- 价值:通过统一接口实现蛋白质序列的 “一站式” 功能解析,去除数据冗余,提升注释效率。
- 应用场景:新发现蛋白质的功能预测(如从宏基因组序列中注释未知蛋白的结构域与功能)、差异表达蛋白的功能富集分析(结合结构域信息揭示核心通路)。
Pfam:蛋白质家族的 “分子指纹库”
- 功能:以多序列比对和隐马尔可夫模型(HMM)构建蛋白质家族,分类为家族、结构域、重复、模体。
- 价值:从序列层面识别蛋白质的保守结构域,为功能预测提供核心依据。
- 应用场景:鉴定蛋白质的功能模块(如激酶的催化结构域)、分析蛋白质家族的进化关系(通过结构域的保守性推断同源性)。
PROSITE:蛋白质功能模式的 “识别器”
- 功能:识别蛋白质序列中的保守域、结构域和功能模体,附带详细的序列、功能、结构描述。
- 价值:从氨基酸序列直接推断蛋白质的功能特征(如酶的活性位点、蛋白互作界面)。
- 应用场景:药物靶点的筛选(识别具有特定功能模体的蛋白质)、突变对蛋白质功能的影响预测(如活性位点突变是否导致功能丧失)。
3. 蛋白质结构分类数据库:结构同源性的 “分类器”
蛋白质结构的分类是理解其进化关系与功能保守性的关键,以下数据库构建了结构分类的 “层级体系”:
CATH:基于结构特征的层级分类
- 分类层级:Class(全 α、全 β、α+β、低二级结构)→ Architecture → Topology → Homologous Superfamily。
- 价值:从结构特征出发,系统归类蛋白质结构域,揭示结构与功能的关联规律。
- 应用场景:未知结构蛋白质的结构类型预测、蛋白质结构进化的宏观分析(如不同物种中同一 Class 结构的分布)。
SCOP:基于进化关系的结构分类
- 分类层级:Class → Fold → Superfamily → Family。
- 价值:更强调蛋白质的进化同源性,通过人工验证确保分类的生物学意义。
- 应用场景:远缘物种蛋白质的进化关系研究(如真核与原核生物中同源结构域的进化分歧)、蛋白质结构创新设计的灵感来源(从不同 Fold 中挖掘结构模块)。

5. AI 驱动的预测结构数据库:突破实验局限的 “新范式”
深度学习的爆发让蛋白质结构预测精度达到实验级别,以下数据库代表了 AI 在结构生物学的革命性进展:
AlphaFoldDB
- 功能:存储 DeepMind AlphaFold 预测的蛋白质三维结构,附带质量评估数据(置信度分数、结构可靠性指标)。
- 价值:填补了实验结构的大量空白(如人类蛋白组中约 60% 的结构此前未被解析),为结构研究提供 “全 coverage” 的资源。
- 应用场景:未解析结构蛋白质的功能预测(如根据 AlphaFold 结构推断其与其他蛋白的互作)、农业物种蛋白组的结构解析(加速作物抗逆机制研究)。
ESM Atlas
- 功能:基于 ESM(进化尺度模型)的语言模型,实现宏基因组蛋白质的高分辨率结构预测,是首个覆盖宏基因组的结构数据库。
- 价值:解析微生物组中大量未知蛋白的结构,为微生物功能研究、新型酶挖掘提供支撑。
- 应用场景:环境微生物中新型降解酶的结构与功能预测、人体肠道微生物组的蛋白质互作网络构建。
4.3.7 互作数据库
在生物信息学研究中,蛋白质互作、遗传互作、病原菌 - 宿主互作等分子间的相互作用是解析生命系统复杂性的核心线索。一系列专业的 “互作数据库” 通过整合实验数据、文献报道和计算预测,为研究者提供了从 “单分子功能” 到 “网络级调控” 的全景视角。
1. 通用蛋白互作数据库:构建分子互作的 “全景网络”
IntAct:蛋白互作的 “实验数据仓库”
IntAct 是专注于蛋白质互作数据存储与共享的数据库,覆盖三类核心互作:
- 蛋白质 - 蛋白质互作;
- 蛋白质 - 核酸互作;
- 蛋白质 - 小分子互作。其数据来源于生物学实验、文献报道和高通量筛选(如酵母双杂交、质谱分析),且详细记录实验方法,为数据可靠性提供溯源依据。
应用场景:
- 新蛋白功能的互作组学验证(如通过 IntAct 查询某蛋白的已知互作伙伴,推测其参与的通路);
- 高通量互作数据的质量评估(对比实验方法与结果的一致性)。
MINT:专家审核的 “高可信度互作库”
MINT 聚焦蛋白质互作的精准注释,所有数据经专家审核且有实验证据支撑。它不仅提供互作关系,还关联蛋白质的分子特性(如基因本体注释、亚细胞定位、结构域),并支持互作网络图生成,直观呈现蛋白间的调控关系。
应用场景:
- 信号通路的精细解析(如通过 MINT 的互作网络,识别某激酶的直接底物与间接调控靶点);
- 蛋白功能的多维度验证(结合结构域与互作信息,推断蛋白的功能模块)。
BioGRID:多维度互作的 “综合资源库”
BioGRID 是覆盖蛋白质、化学、遗传互作的综合性数据库,数据来源于多物种、多实验方法(实验室实验、文献、高通量筛选)。它注重数据质量与验证,提供数据筛选和质控步骤,确保信息的可靠性。
应用场景:
- 跨物种互作的比较分析(如人类与小鼠中某通路互作的保守性研究);
- 药物 - 靶点互作的初筛(从化学互作数据中识别潜在的小分子调控靶点)。
STRING:种内互作的 “预测 + 实验” 双引擎
STRING 专注于种内蛋白质互作,覆盖人类、小鼠、线虫等常见模式生物及稀有物种。它通过 “实验验证 + 计算预测” 双重手段确保数据准确性,且提供强大的可视化与分析工具(序列比对、功能注释、网络分析、富集分析)。
应用场景:
- 基因集的功能富集与互作网络构建(如差异表达基因的互作网络分析,揭示核心调控模块);
- 未知蛋白的功能预测(通过 STRING 的同源互作数据,推断新蛋白的潜在功能)。
2. 整合型与专项互作数据库:从通路到病原菌 - 宿主互作的 “深度解析”

