感知上下文并可解释地预测合成致死药物靶点的大语言模型研究
仅供个人备忘使用,如果涉及到其他问题,请联系我删除。
1 合成致死概念解析,老生常谈。
2 合成致死是抗癌药物靶点金矿,敲带有癌症特异性突变的SL伴侣基因将杀死癌细胞,但不会影响正常细胞生存。
3 湿实验筛选时间长成本高(大规模数据和生物学机制)
4 深度学习计算方法的深度学习方法:基于图神经网络的方法(GNNs),问题建模:SL预测问题→链接预测 ,在图上进行图表征学习,节点对应的是基因,在节点上进行表征学习,对边进行表征学习进行下游预测,
5 预测方法:上下文无关方法:为基因生成通用表征进行SL预测,上下文特异性方法:基于特定癌症类型或细胞系,生成上下文特定的基因表示来预测SL;
6 数据稀疏性:上下文特异性SL预测方法的主要挑战,只有31种细胞系具有SL数据标签,已有标签数据的基因覆盖率也很低
7 限制SL方法泛化能力弱,不同细胞系SL关系可能逆转
8 其他挑战:缺乏可解释性,正负样本不平衡,基因对的长尾分布
9 基于Transformer的与训练大语言模型:
生物大语言模型:蛋白质:ESM;单细胞组学语言模型:Geneformer,scBERT,scGPT,scFOUNDATION
通用大预言模型:GPT,DeepSeek
LLM的优势:更少依赖标签数据,具有小样本学习能力,有更强的泛化和迁移能力,可生成自然语言解释
大语言模型的四个方法:
Mit4SL:在基因表征学习的基础上,显式加入细胞系的表征,基因表征和细胞系表征的解耦,让模型能适应新的细胞系实现跨细胞系的预测。
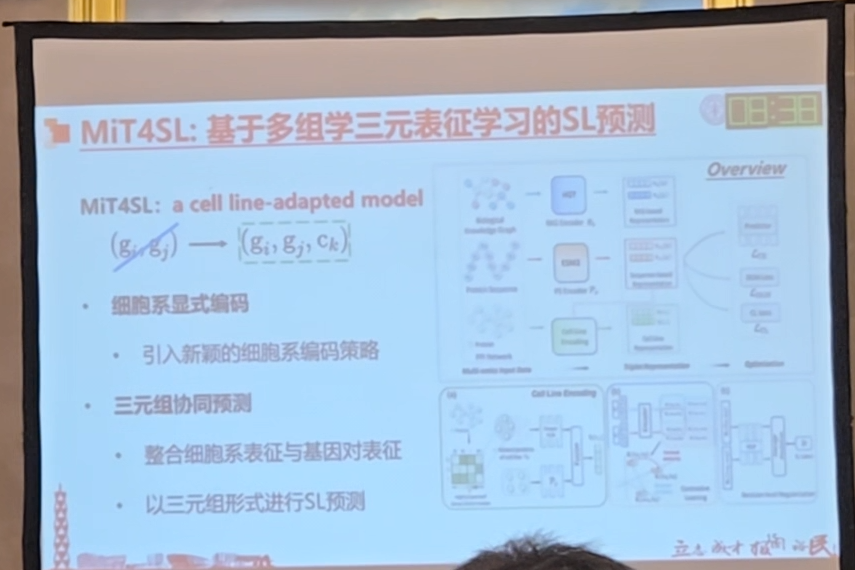
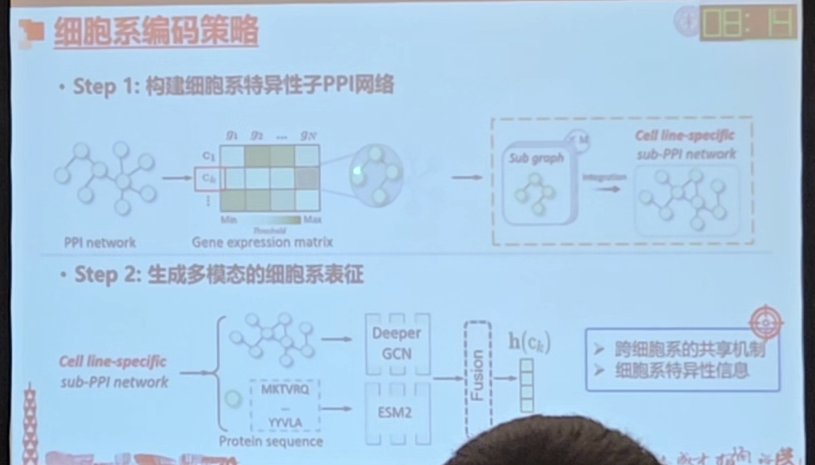
采用转录组数据和蛋白质-蛋白质相互作用网络,根据基因表达量通过阈值去筛选某一个细胞系中特异性PPI子网络,用子网络代替细胞系,
从子网络抽取蛋白质,把序列投入蛋白质语言模型中,根据序列生成细胞系的表征,之后融合在一起,代表细胞系
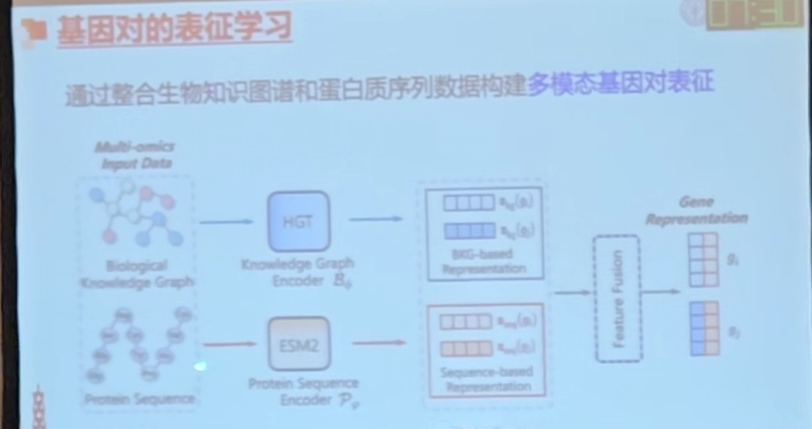
另外,表征了基因通过知识图谱的学习和ESM2生成基因的表征,获得三元组的表征
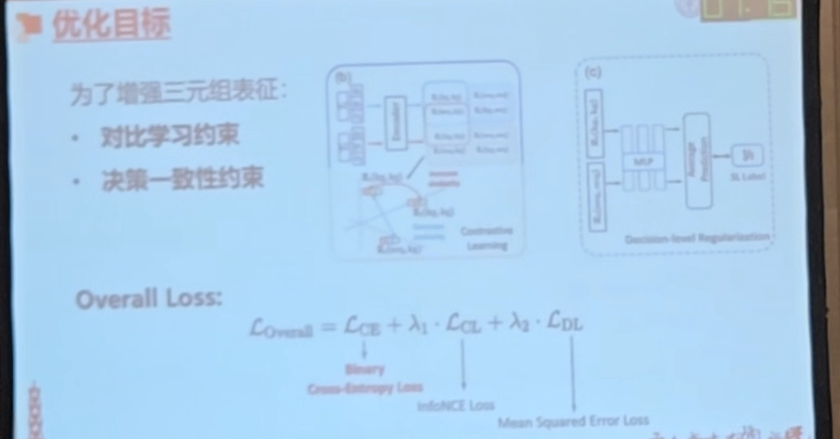
为了强化三元组表征,引入对比学习和决策一致性约束,三个损失
MIT4SL在不同细胞系获得改善
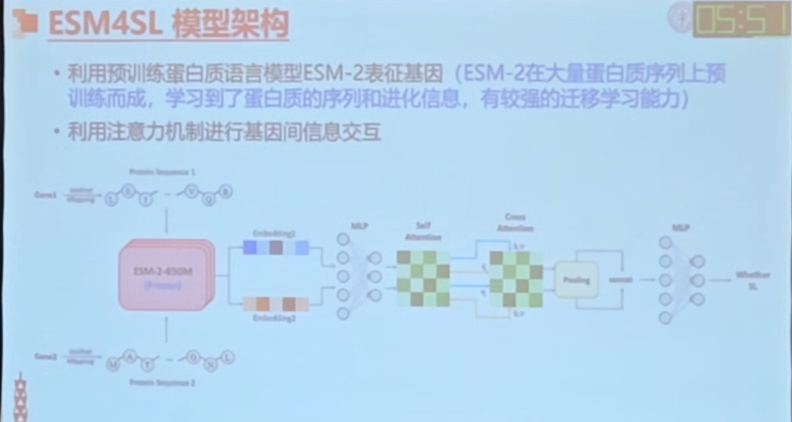
ESM4SL
出发点针对长伟基因,泛化能力不强,关键假设是合成致死本质是两个基因或蛋白质之间的功能冗余性,用大语言模型捕捉蛋白质功能间的关系。

通过进化和序列信息推到功能关系,把两个蛋白质扔到ESM2里面生成表征,然后通过交叉注意力机制,进行有监督学习,发现提升长尾场景能力。
PromptCASL
基于组学和自然语言的大语言模型,框架包含两个部分,左边是上下文特异性特征的提取,右边是上下文无关的特征的提取,左边把基因和细胞系的信息放到提示词的模板里面再交给biomedBERT大语言模型生成表征,组学数据也通过细胞系语言模型生成表征,右边通过知识图谱基因PT自然语言的大语言模型,生成基因的表征上下文无关的;然后把特征融合交给下游训练。

在多细胞系训练,全新细胞系预测提升明显,发现注意力机制从标点符号与结构标记转机到具有生物学意义的词元,说明能抓住生物学意义
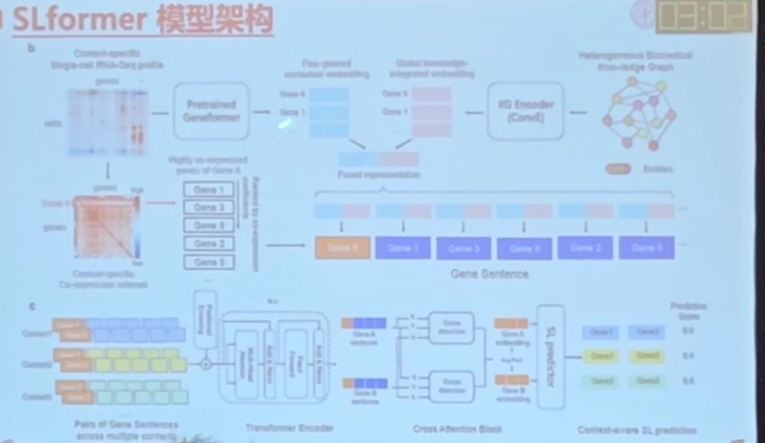
SLformer
基于单细胞数据的交给基因former来生成对应的表征,结合知识图谱的表征结合到一起,生成基因sentence,抓住基因的上下文,有湿实验
总结展望
为了应对数据稀疏:
使用预训练大语言模型,大语言模型有小样本学习、泛化、迁移能力;Transformer具有上下文感知的优势,注意力机制和基因表征能提供一定的可解释性;
未来探索方向:
融合更多组学数据(蛋白质组、代谢组、表观基因组)
SL上下文特异性与耐药性的关系
结合单细胞扰动效应预测
从细胞系拓展到类器官模型
预测SL靶点的可成药性和毒性
