模拟数据生成---使用NGS数据模拟软件VarBen
目录
1.在BAM文件中根据指定的变异等位基因分数的指定位置或区域随机选择read。
2.筛选变异等位基因分数的reads:
3.装BWA和samtools软件包(samtools在linux系统中下载过,前文有讲过)
4.写py脚本
5.下载pysam库模块
6.下载参考基因组hg38
7.解压gz
8.建立samtools索引
9.建立BWA索引
10.下载picard工具包
11.对hg38.fa建立索引
12.使用BWA的index命令为参考基因组文件创建索引
13.BWA进行比对
14.对bam文件进行标记重复并移除重复的读取
15.使用变体检测工具:运行GATK、FreeBayes、Samtools等变体检测工具,在比对后的数据中检测SNV和Indel,并生成VCF文件。
16.下载transverse colon的wgs的vcf文件
编辑
17.尖峰点突变(SNV 和 InDel)进入 bam 文件。(Illumina平台)
18、变异检测验证(FreeBayes / GATK / samtools)
19、Tips 与建议
浅浅介绍一下VarBen是什么东东吧。
VarBen是用于向bam文件添加变异模拟的工具,包括单核苷酸变异、短的插入和缺失以及结构变异。
VarBen主要在linux系统下使用,无需编译即可运行。但是需要预先配置运行环境。
采用的是比对到参考基因组特定位点的测序reads进行编辑的方式来进行模拟。这种方法可保留测序过程中产生的错误类型分布模式,使生成的模拟数据更接近实际数据。那么我们应该怎么做呢?请听我一本正经的胡说八道吧~
1.在BAM文件中根据指定的变异等位基因分数的指定位置或区域随机选择read。
【概念:等位基因分数是一个用于描述在某个基因座中,某个特定等位基因出现的频率或比例的指标。】
首先,确保你的BAM文件已经被索引。你可以使用samtools index命令为BAM文件创建索引。这个索引文件(.bai)将允许你快速查询BAM文件中的特定区域。
(具体操作在前文有讲过哦~)

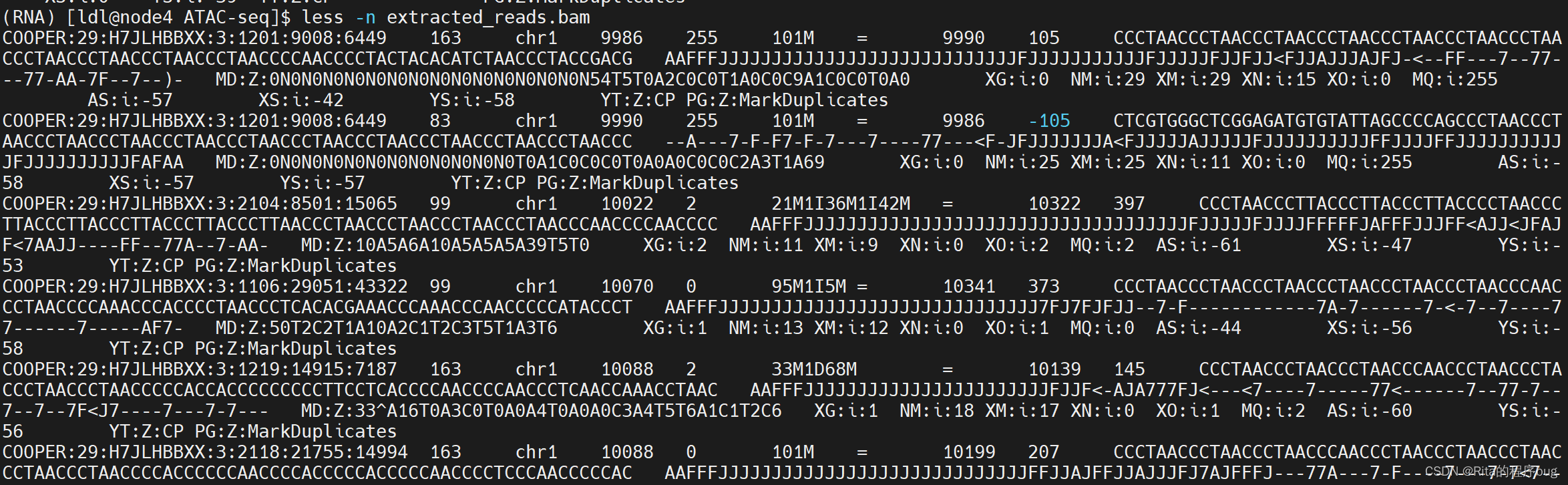
利用samtools view命令,你可以提取指定区域或位置的reads(提取染色体chr1上位置10000到20000之间的reads)。
samtools view sorted_ENCFF845HFA.bam chr1:10000-20000 >extracted_reads.bam
查看文件:
less -n extracted_reads.bam
2.筛选变异等位基因分数的reads:
这一步通常需要使用一些自定义的脚本或工具,因为标准的SAMtools并不直接支持根据等位基因分数筛选reads。你可以使用Python的pysam库或者其他编程语言的相关库来读取BAM文件,并筛选出具有特定等位基因分数的reads。
大家放心,在Linux中使用Python是非常简单的,因为大多数Linux发行版都预装了Python。
#检查python版本
python --version

(我的是python3版本,你的是什么版本呢?)
3.装BWA和samtools软件包(samtools在linux系统中下载过,前文有讲过)
#如果你也是python3版本的话
pip3 install BWA
#如果不是
pip install BWA下载完成标志:
4.写py脚本
我在linux专栏中有讲到vim文本编辑器,可以用来编辑python脚本的。除此之外,你还可以使用任何文本编辑器(如nano、emacs、gedit等)来编写Python代码,并将其保存为.py文件。然后,你可以使用上述方法在终端中运行它。
vim reads.py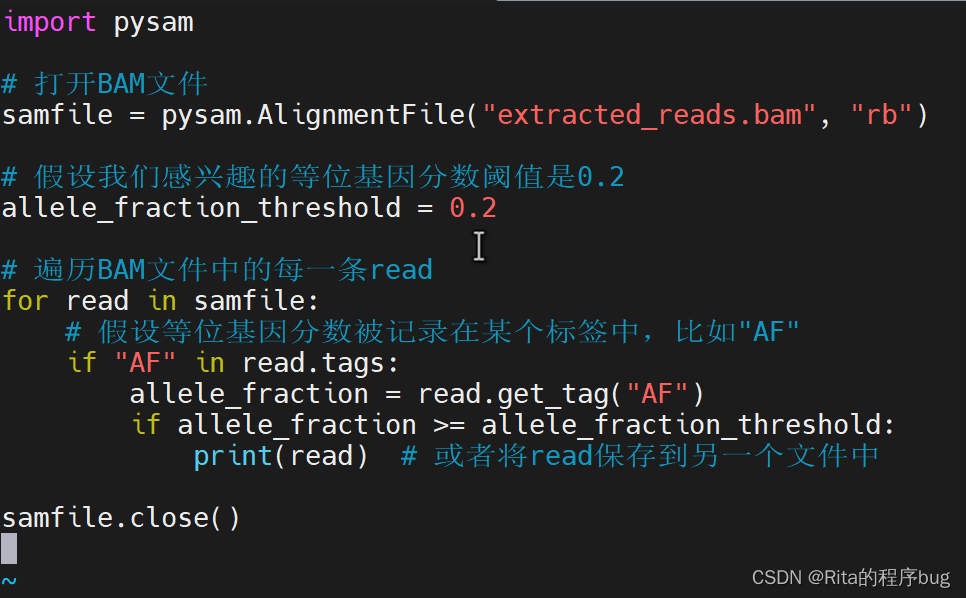
vim命令:
i进入编辑模式
Esc退出编辑
:wq保存并退出
【忘记的话可以回去看看我之前写的文章(linux专栏)哦】
5.下载pysam库模块
pip3 install pysam我一开始以为我以为python3会自带的,一运行脚本发现报错,原来是我自作多情了,所以如果你也没有安装的话,需要安装一下呢。(以下为报错信息)
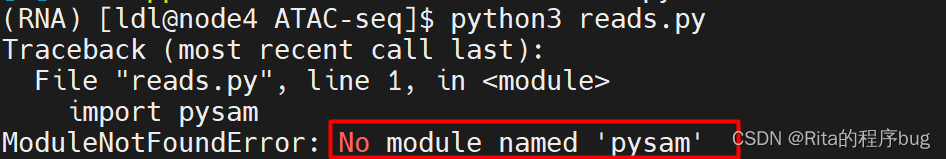
【报错是常态】
果然,我又再一次报错了。
尝试第二种方法:通过conda来安装pysam
conda install -c bioconda pysam6.下载参考基因组hg38
下载UCSC工具包
conda insatll UCSC进入encode官网
https://genome.ucsc.edu/![]() https://genome.ucsc.edu/
https://genome.ucsc.edu/
选择Genome Data

复制下载地址后,用wget下载
wget -b https://hgdownload.soe.ucsc.edu/goldenPath/hg38/bigZips/analysisSet/hg38.analysisSet.2bit
7.解压gz
gunzip hg38.fa.gz8.建立samtools索引
samtools faidx hg38.fa建立索引后会多一个fai后缀的文件
9.建立BWA索引
bwa index -a bwtsw hg38.fa10.下载picard工具包
wget https://github.com/broadinstitute/picard/releases/download/2.23.0/picard.jar11.对hg38.fa建立索引
java -jar picard.jar CreateSequenceDictionary R=hg38.fa O=hg38.dict
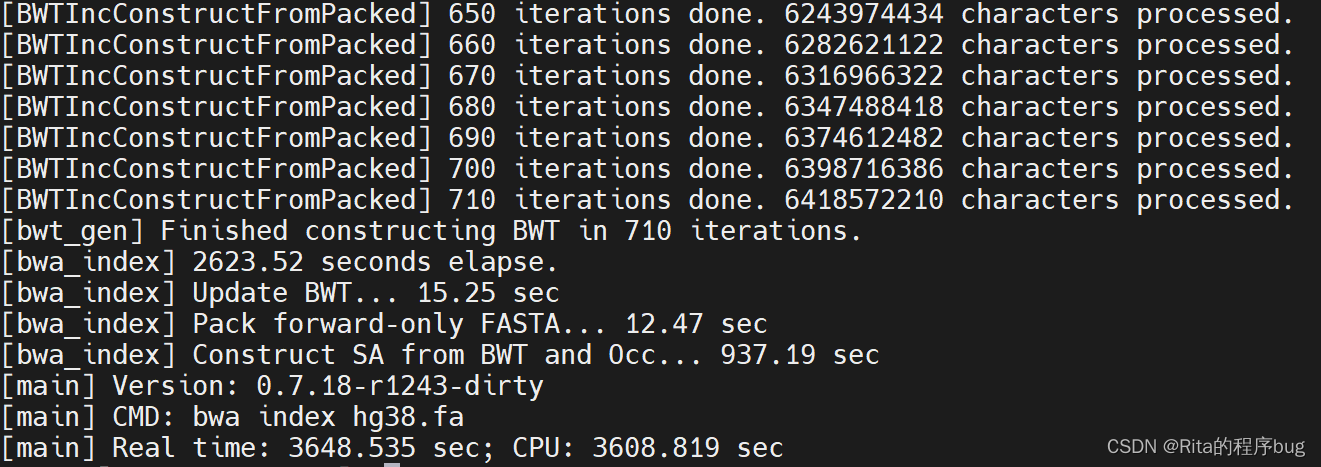
12.使用BWA的index命令为参考基因组文件创建索引
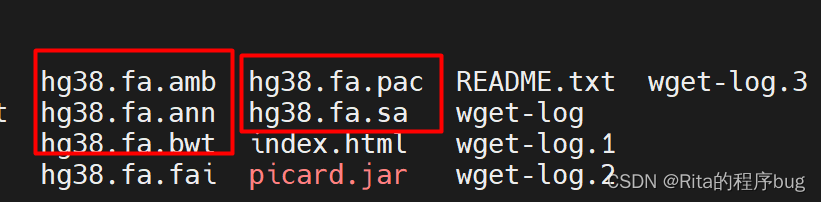
bwa index hg38.fasta这将在当前目录下生成一系列以参考基因组文件名为前缀的索引文件(如hg38.fasta.amb, hg38.fasta.ann, hg38.fasta.bwt, hg38.fasta.pac, hg38.fasta.sa)。
13.BWA进行比对
BWA (Burrows-Wheeler Aligner) 是一个常用的短序列比对工具,但它并不直接对BAM格式的文件进行比对。BAM是Binary Alignment/Map的缩写,是SAM (Sequence Alignment/Map) 格式的二进制版本,用于存储序列比对的结果。
14.对bam文件进行标记重复并移除重复的读取
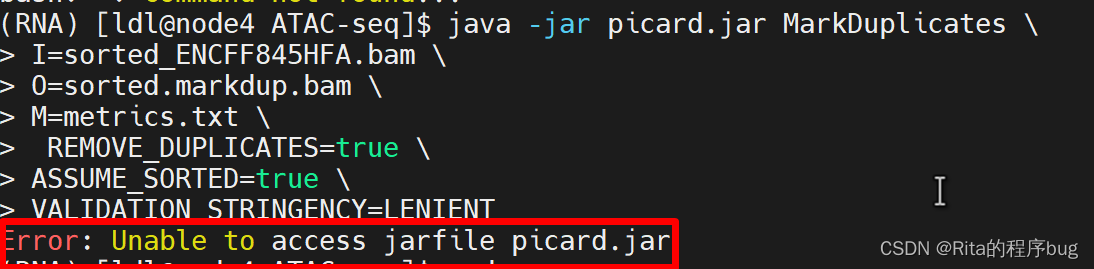
java -jar picard.jar MarkDuplicates \ I=sorted_ENCFF845HFA.bam \O=sorted.markdup.bam \ M=metrics.txt \ REMOVE_DUPLICATES=true \ ASSUME_SORTED=true \ VALIDATION_STRINGENCY=LENIENTI指定输入BAM文件。O指定输出BAM文件,其中重复的读取将被标记或移除(取决于REMOVE_DUPLICATES参数的值)。M指定一个输出文件,用于保存关于重复标记的统计信息。REMOVE_DUPLICATES设置为true将会从输出BAM文件中移除重复的读取;如果设置为false,则只会标记它们但不会移除。ASSUME_SORTED设置为true表示输入BAM文件已经排序(这是必须的,因为MarkDuplicates需要排序的输入)。VALIDATION_STRINGENCY设置为LENIENT表示在读取BAM文件时采用宽松的错误检查。
【注意,这里的代码你需要改的是I=sorted_ENCFF845HFA.bam这个,这个为你自己的文件】
【另外,也要确保picard.jar在你当前的目录下,不然你就会收到一条报错信息。】
Error: Unable to access jarfile picard.jar
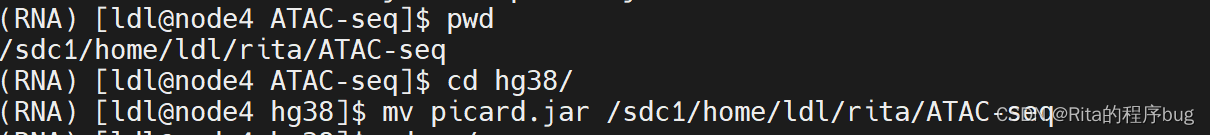
于是,我进行了以下操作
好了,回归正题,标重的页面是怎样的呢
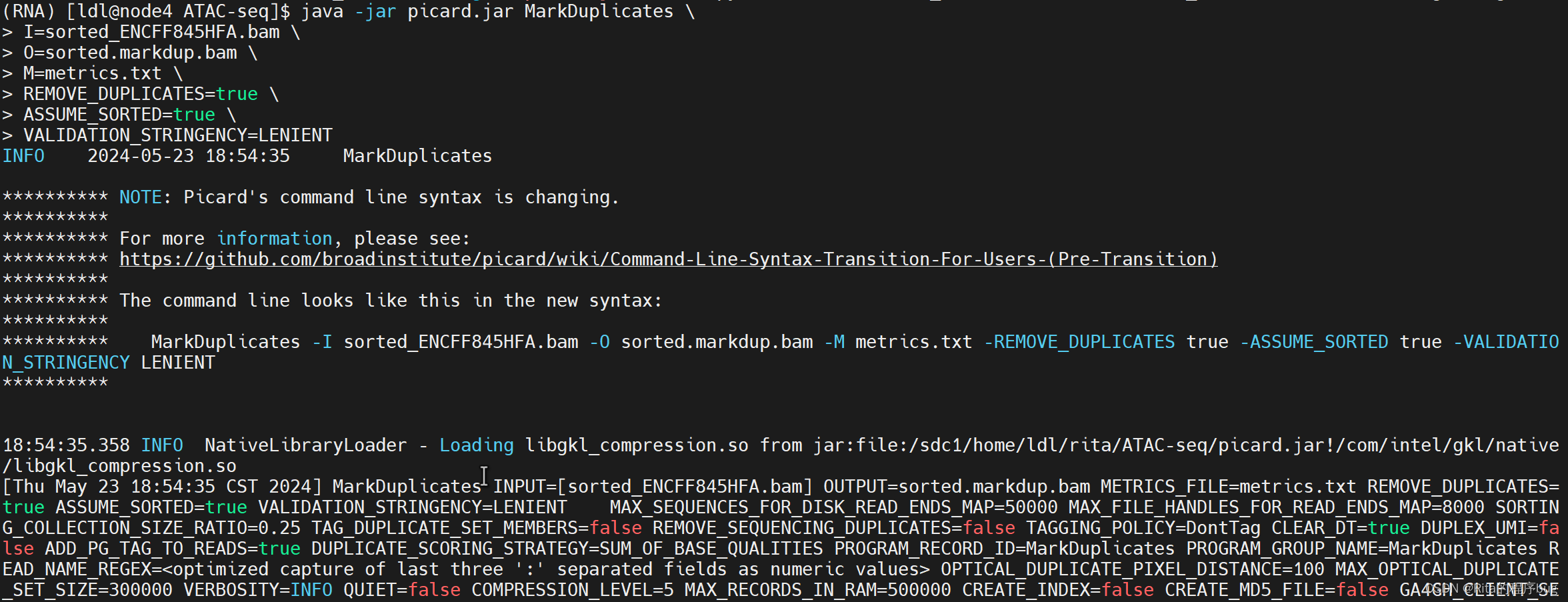
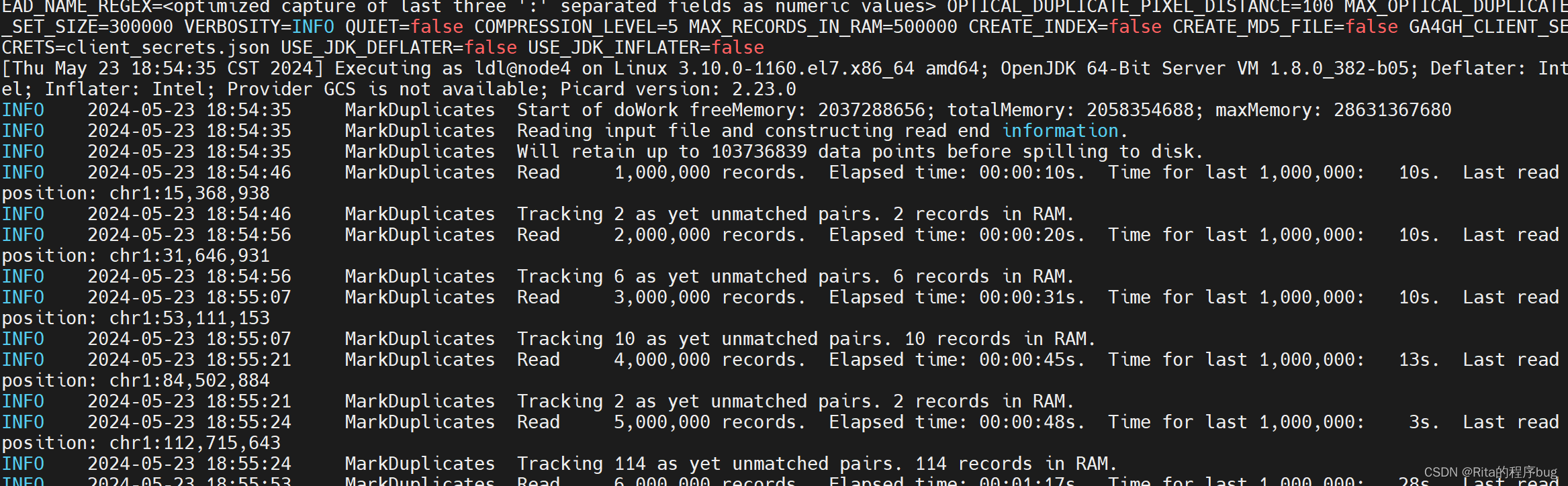 (这个时候是需要一点等待的时间的)
(这个时候是需要一点等待的时间的)
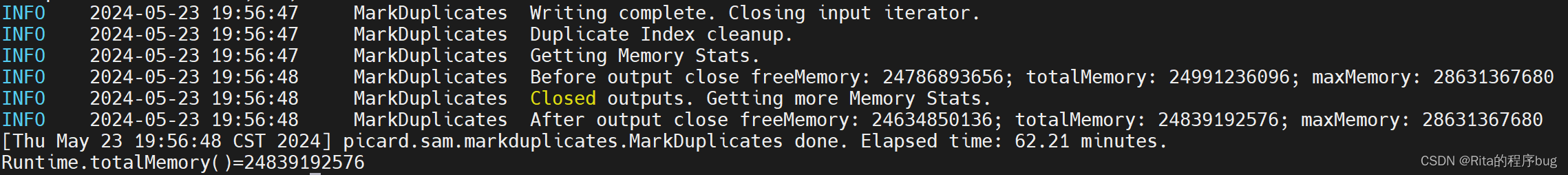
(跑了一个多小时,终于跑完)
15.使用变体检测工具:运行GATK、FreeBayes、Samtools等变体检测工具,在比对后的数据中检测SNV和Indel,并生成VCF文件。
#下载bcftools
conda install bcftoolsbcftools是用于从一个排序并标记了重复的BAM文件中生成原始的单核苷酸变异(SNV)和小的插入/删除(indel)位点。
samtools mpileup -uf hg38.fa sorted.markdup.bam | bcftools view -Obvcg - > variants.raw.bcfmpileup是samtools的一个工具,用于生成基因型似然性(pileup)格式的输出,它表示了给定参考基因组上每个位置的测序读数覆盖和碱基质量。-f gh38.fa:指定参考基因组文件(上面在encode下载下来那个)- sorted.markdup.bam:输入文件,这是一个已排序且标记了重复的BAM文件。
bcftools是处理Variant Call Format (VCF) 和 Binary Variant Call Format (BCF) 文件的工具集。view子命令用于查看、转换和过滤这些文件。

16.下载transverse colon的wgs的vcf文件
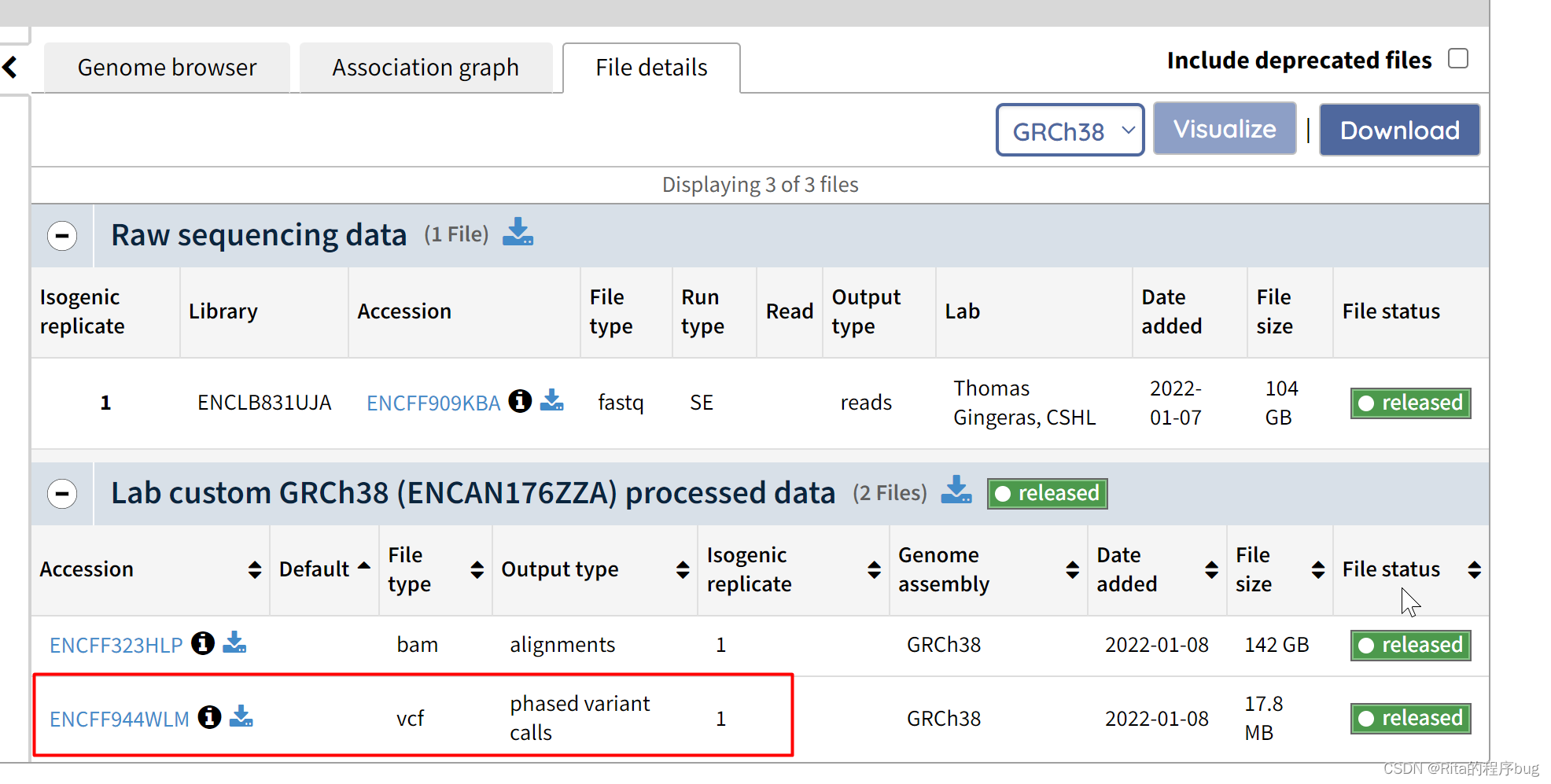
17.尖峰点突变(SNV 和 InDel)进入 bam 文件。(Illumina平台)
GitHub - nccl-jmli/VarBen
18、变异检测验证(FreeBayes / GATK / samtools)
GATK HaplotypeCaller 示例:
gatk HaplotypeCaller \-R Homo_sapiens.GRCh38.dna.primary_assembly.fa \-I mutated_dedup.bam \-O final_calls.vcf.gz
对比一下你注入的变异和检测出来的结果,是不是神还原?
19、Tips 与建议
如果你是科研模拟验证算法,可以用 VarBen 插入一组已知变异,跑一套变异检测 pipeline 看检出率。
如果你是教学使用,可以插入多种变异(SNP/InDel/结构变异)并配合 IGV 展示。